I marcatori molecolari, sequenze di DNA genomico associate ad un locus genico di interesse, rappresentano uno strumento indispensabile per:
1 promuovere l'innovazione varietale nel settore del miglioramento genetico della canapa così come è stato per le colture di interesse agrario
2 le esigenze di tipizzazione univoca di una varietà (fingerprinting) e la tutela dei diritti dei costitutori
3 il riconoscimento delle varietà da droga da quelle da fibra.
Una delle maggiori difficoltà nella messa a punto di questi tool in canapa risiede nell'elevata eterogeneità intra-varietale che è tipica di questa specie a comportamento allogamo. La disponibilità sempre crescente di dati di sequenziamento del genoma (piattaforme NGS) consente di individuare migliaia di polimorfismi (SNP, InDel) su tutto il genoma e di poter sfruttare questi siti variabili per la costituzione di marcatori molecolari high throughput la cui analisi sui materiali genetici può essere automatizzata.
 Cannabis sativa L.: un'officina naturale di biomolecole e prodotti derivati dalla coltivazione dai molteplici utilizzi
Cannabis sativa L.: un'officina naturale di biomolecole e prodotti derivati dalla coltivazione dai molteplici utilizzi
Nell'ambito del progetto UNIHEMP, si sono resi disponibili i dati di sequenziamento del genoma di 4 varietà di canapa su polimorfismi di tipo SNP ed InDel sia a livello intergenico che nelle regioni codificanti per geni coinvolti nella sintesi dei tre metabolismi di interesse (cannabinoidi, terpeni e flavonoidi).
E' stato quindi possibile sviluppare un protocollo per analisi High Resolution Melt (HRM) in canapa a partire dalla validazione dei polimorfismi individuati in geni già noti. L’HRM è una metodica di analisi high throughput post amplificazione PCR che permette di individuare/validare variazioni genetiche in una sequenza di DNA.
La tecnologia sfrutta la dissociazione del dsDNA all'aumentare della temperatura (curva di melting o di dissociazione). Il saggio HRM è stato messo a punto su due strumenti e relativi software di analisi: QS3 Real-Time PCR System e Rotor-Gene 6000. Il primo passaggio ha previsto un'attenta progettazione del set di primers da utilizzare dal momento che la lunghezza degli ampliconi incide sulla sensibilità e specificità dell’analisi HRM in misura inversamente proporzionale per cui maggiore è la dimensione degli ampliconi e minore sarà la capacità discriminante del saggio.
Per ragioni riservatezza dei dati non ancora oggetto di pubblicazione, non si rendono note le sequenze dei marcatori sviluppati con questa tecnologia. Per informazioni si prega di inviare una mail a laura.bassolino@crea.gov.it e roberta.paris@crea.gov.it
Ad oggi non risultano lavori scientifici pubblicati sulla tecnologia HRM in canapa, sebbene è pensabile che le aziende floro-vivaistiche siano già in possesso della tecnologia per le finalità del breeding per lo sviluppo di nuove varietà, pertanto l'innovazione tecnologica proposta dal CINLab può da un lato colmare un gap scientifico nel settore del miglioramento genetico in questa specie e dall'altro offrire uno strumento ai costitutori per tipizzare le varietà loro prodotte e quindi tutelarle sul mercato.
La metodica di analisi HRM ha diversi ambiti di applicazione che vanno dalla genotipizzazione basata su marcatori molecolari al miglioramento genetico all'identificazione di contaminanti a livello di sementi.
I marcatori sviluppati dal CINLab su sequenze SNPs ed InDels consentono di discriminare le varietà per chemotipo e a seguito di ulteriore sviluppo e validazione di distinguere varietà diversamente pigmentate.
 Sviluppo di marcatori molecolari per la tipizzazione varietale in canapa
Sviluppo di marcatori molecolari per la tipizzazione varietale in canapa
L'innovazione tecnologica proposta ha diversi ambiti di applicazione, tra questi:
- Breeding assistito da marcatori (MAS)
- Fingerprinting varietale
- Finalità forensi: identificazione varietà da droga
Il CINLab ha sviluppato nell'ambito delle risorse del progetto UNIHEMP dei marcatori molecolari basati su polimorfismi di tipo SNP e InDel su sequenze geniche codificanti per enzimi noti per i pathways metabolici deputati alla sintesi di metaboliti secondari di interesse in canapa per le loro diverse applicazioni industriali quali cannabinoidi, terpeni e flavonoidi.
I marcatori molecolari sono stati sviluppati per analisi di High Resolution Melting (HRM) ed il saggio messo a punto su strumenti quali QS3 Real-Time PCR System e Rotor-Gene 6000 e relativi software di analisi dei dati.
Per motivi di riservatezza in attesa di pubblicazione, le sequenze nonché l'indicazione della posizione dei polimorfismi sul genoma non viene resa nota in questa fase.
La tecnologia è già stata validata in laboratorio (TRL 4) sebbene su un numero limitato di genotipi, pertanto, un'ulteriore fase di ottimizzazione è richiesta nei prossimi mesi con l'obiettivo di sviluppare un numero sempre maggiore di marcatori molecolari per le finalità del breeding e della tutela varietale.
Prof. Enrico Francia, Laboratorio BIOGEST accreditato alla rete. La collaborazione si è sviluppata nell'ambito del progetto PON ARS01_00668 UNIHEMP “Utilizzo di biomassa da canapa industriale per la produzione di energia e nuovi biochemicals” (Prot. n. 2016 del 27/07/2018)
La tecnologia è stata validata su alcuni genotipi di canapa già sequenziati ed è in fase di validazione su un numero più ampio di materiali al fine di testare l'effettiva robustezza dei marcatori.
I dati non sono stati ancora rilasciati come report scientifici (pubblicazioni, atti convegno) ma questi sono in preparazione e, completata la fase di validazione, verranno rilasciati.
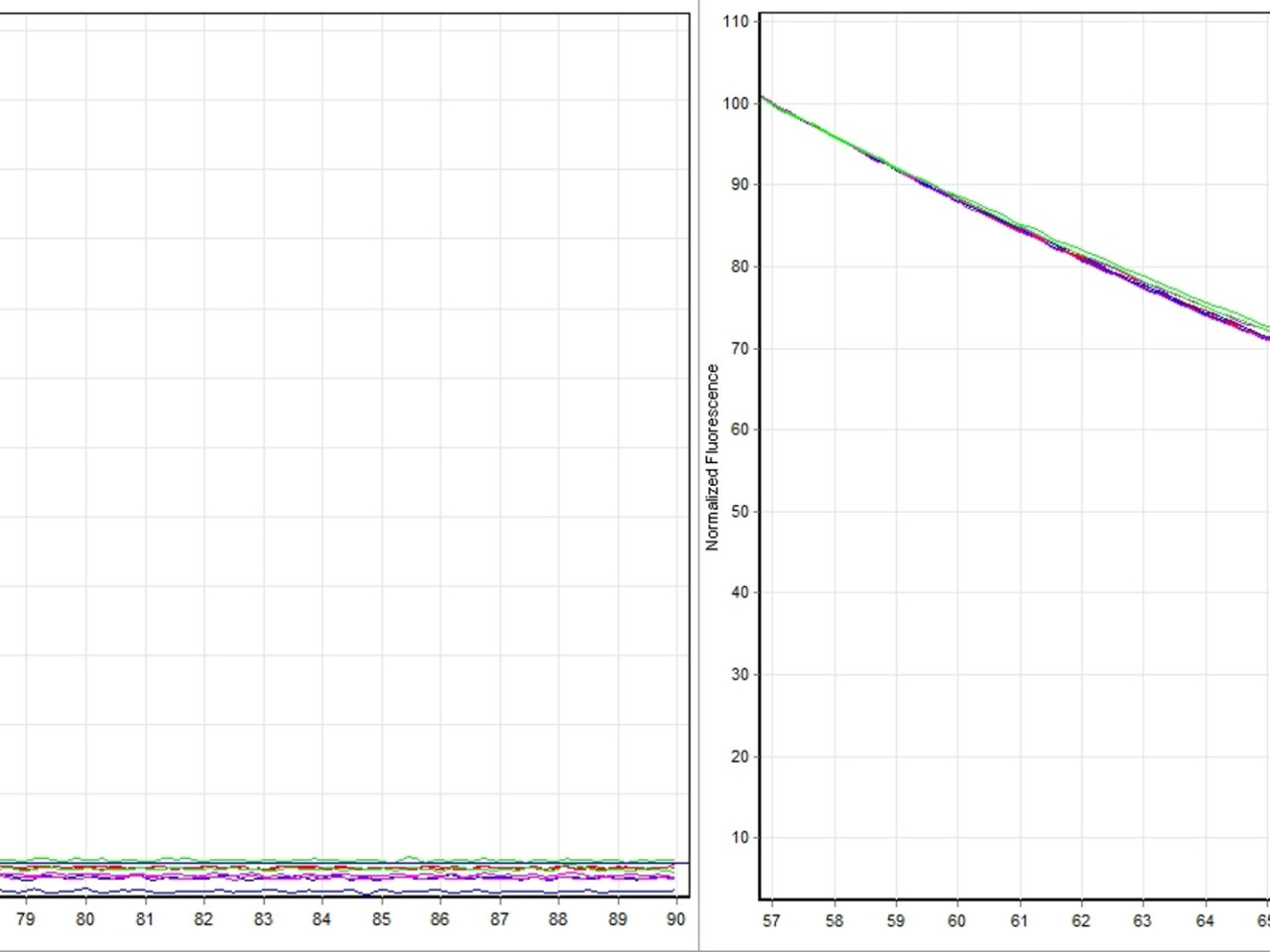 Esempio di un profilo di melting per un marcatore tipo InDel che consente di discriminare tre diversi genotipi
Esempio di un profilo di melting per un marcatore tipo InDel che consente di discriminare tre diversi genotipi

